Anna Marzucco et al. Оценка эффективности нового диагностического теста для прямого обнаружения РНК SARS-CoV-2 с использованием портативной системы MOLECULAR MOUSE.
Предпосылки
Диагностика COVID-19 в значительной степени зависит от молекулярных анализов с коротким сроком выполнения. Большинство диагностических наборов для выявления генома SARS-CoV-2 требует наличия квалифицированного персонала, большого количества приборов и рабочих процессов, которые могут быть выполнены только в высокоспециализированных лабораториях, что подразумевает высокую стоимость и задержки в проведении теста.
Мы оценили клиническую эффективность теста MOLECULAR MOUSE (MM) DIRECT COVID-19 с помощью системы MOLECULAR MOUSE, инновационного миниатюрной платформы для ПЦР в реальном времени, для выявления РНК SARS-CoV-2 непосредственно из оро/назофарингеальных мазков примерно за один час. Тест состоит из раствора для лизиса раствора для простой подготовки образца и готового к использованию картриджа Lab-On-Chip содержащий все реагенты для специфического обнаружения вирусного ORF1b и гена нуклеокапсидного и гена нуклеокапсидного белка (N1 и N2).
Целью исследования была оценка ММ DIRECT COVID-19 в сравнении с клинико-диагностическими характеристиками AllplexTM SARS-CoV-2 (Seegene), референсным методом RT-PCR, который регулярно используется в нашей лаборатории.
Методы
Оценивались 689 свежих и замороженных оро/назофарингеальных мазков (собранных в течение 48 ч от начала симптомов) положительных и отрицательных пациентов с COVID-19. Образцы были собраны в период с 23/08/2021 по 07/10/2021. Анонимизированные образцы, отобранные с различной вирусной нагрузкой, были проанализированы одновременно с помощью MM DIRECT COVID-19 и референсным методом. Остаточные образцы мазков загружались на платформу MOLECULAR MOUSE для валидационного исследования в соответствии с инструкциями производителя.
Результаты
Результаты теста MM DIRECT COVID-19 на 689 образцах (247 положительных и 442 отрицательных на Allplex SARS-CoV-2), показали 94,33% чувствительности (233/247) и 99,32% специфичности (439/442) по сравнению с референсным методом (табл. 1). Все 14 ложноотрицательных результатов были получены при высоком значении Ct (между 30-36), что указывает на низкую чувствительность теста только в образцах с низкой вирусной нагрузкой.
Выводы
Мы продемонстрировали, что тест MM DIRECT COVID-19 является точным для подтверждении диагноза SARS-CoV-2. Даже менее опытные пользователи могут выполнить его
простой рабочий процесс и получить результаты примерно за один час. В заключение следует отметить, что мы провели клиническое обоснование быстрого, специфичного и простого в использовании теста, который может способствовать децентрализации тестирования на SARS-CoV-2 в небольших неспециализированных подразделениях (т.е. в клиниках неотложной помощи), в том числе в развивающихся странах.
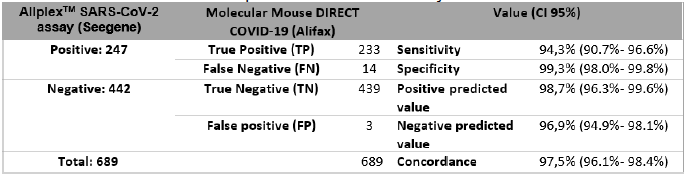
Источник: Постерная сессия на конференции ECCMID 2022, Лиссабон, Португалия.
Vittorio IvagneIvagnes et al. Прямое обнаружение генов резистентности к ß-лактамам из положительной культуры крови с помощью чипа для определения резистентности к грамотрицательным бактериям (система Molecular Mouse).
Инфекция кровотока (ИК) - серьезное заболевание, которое может привести к генерализованной инфекции, множественной органной недостаточности и смерти. Быстрая идентификация возбудителя инфекции в положительных культурах крови (КК) (и их возможных маркеров резистентности) имеет решающее значение для своевременного начала эффективной антибиотикотерапии.
По данным Европейского центра профилактики и контроля заболеваний, в 2021 году наиболее резистентными к одному или нескольким классам антибиотиков были следующие виды бактерий: E. coli (39,4%), K. pneumoniae (11,9%). β Лактамы являются одними из наиболее используемых препаратов для лечения ИК, вызванных грамотрицательными бактериями, однако резистентность к этим антимикробным препаратам выросла за последние десятилетия, представляя собой всемирную клиническую проблему. Различные коммерческие тесты нуклеиновых кислот для диагностики ИК могут сократить время идентификации генов резистентности и имеют дополнительную ценность для принятия решений о рациональном использовании антибиотиков, сокращая время до начала соответствующей и оптимальной антибиотикотерапии.
Мы оценили эффективность платформы Molecular Mouse (производитель Alifax) с чипом для определения резистентности грамотрицательных бактерий (GNR) для выявления 9 генов резистентности к β-лактамазам blaIMP, blaCMY 2, группа blaCTX M 1\9, blaKPC, blaNDM, blaOXA-23 подобные, blaOXA-48 подобные, расширенные бета-лактамазы blaSHV, blaVIM) непосредственно из положительной культуры крови (ПКК).
Методы
Для оценки чипа GNR мы использовали 4 вида бактерий (K. pneumoniae, P. aeruginosa, E. coli, A. baumannii) с общим количеством 60 образцов и 74 генами резистентности. Мы использовали архивные образцы из положительных КК на вышеупомянутые гены резистентности по 1,5 мл, смешанные со 100 мкл диметилсульфоксида и хранившиеся при 80 С, которые были предварительно исследованы с помощью системы BacTAlert (bioMérieux).
Кроме того, мы дополнили исследование положительными симулированными КК, чтобы увеличить количество генов резистентности для анализа. Для получения симулированных КК были взяты свежие колонии грамотрицательных бактерий, выросшие на чашках с агаром Макконки и суспендированные в фосфатном забуференном солевом растворе и инокулированы во флаконы КК с 8 мл консервированной человеческой крови. Инокулированные флаконы были инкубированы в системе BacTAlert до получения положительного сигнала. 200 мкл бульона положительных КК анализировали с помощью чипа GNR в соответствии с инструкциями производителя и результаты были получены через 1 ч. Результаты анализа GNR сравнивали с результатами ПЦР-амплификации и секвенирования субкультивированных изолятов в качестве референсного метода.
Результаты
Всего в исследование было включено 58 положительных КК (56 мономикробных и 2 полимикробных). Анализ GNR правильно идентифицировал 74 (100%) из 74 β-лактамаз из 60 микроорганизмов, включая CMY (n=10), CTX M (n=16), KPC (n=13), NDM (n=9), OXA 23 (n=14), OXA 48 (n=5), ESBL SHV, (n=1), IMP (n=1) и VIM (n=5).
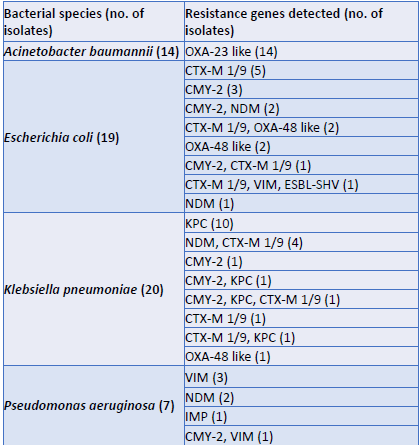
Таблица 1. Профили генов антимикробной резистентности видов микроорганизмов включенных в исследование.
Выводы
Эти предварительные данные свидетельствуют о потенциальной целесообразности использования чипа GNR в качестве полезной альтернативы фенотипическому тестированию для быстрого выявления маркеров резистентности и могут стать важным подспорьем в оптимальном ведении пациентов в критическом состоянии. Необходимо увеличить количество образцов и провести дальнейшие исследования для более точной оценки эффективности.
Источник: Анналы Università Cattolica del Sacro Cuore Rome (Italy), 2022.
Vittorio Ivagnes et al. Быстрая идентификация видов Candida из положительных культур крови с помощью молекулярного анализа на картридже lab-on-chip.
Задачи:
Кандидемия, в частности связанная с неальбикансными видами Candida, является причиной значительной заболеваемости и смертности, а также увеличения расходов на здравоохранение, связанных с длительным пребыванием в стационаре. В связи с продолжающейся тенденцией к увеличению числа вспышек Candida auris и C. parapsilosis своевременная идентификация видов Candida в положительных культурах крови приобретает решающее значение для ведения пациентов и остается сложной задачей.
Целью настоящего исследования была оценка недавно разработанной платформы Molecular Mouse (производитель Alifax S.r.l.) на чипе в режиме реального времени для выявления клинически значимых видов Candida непосредственно из положительных культур крови.
Материалы и методы:
Мы оценили анализ на чипе Yeast Blood (YBL) в сочетании с собственным протоколом выделения ДНК на 106 положительных культурах крови (КК) (63 симулированных и 43 реальных КК). В частности, сначала анализ был проведен на 7 смоделированных КК, включающих каждый из следующих видов Candida: C. albicans, C. glabrata, C. parapsilosis, C. tropicalis, C. krusei, C. auris, C. guillermondii, C. lusitaniae и C. dubliniensis. Затем анализ был проведен на 43 реальных положительных КК и сравнен с результатами, полученными с помощью MALDI-TOF-идентификации.
Результаты:
Молекулярный анализ на чипе в режиме реального времени позволил нам идентифицировать все виды Candida, инокулированные в симулированных положительных КК, за исключением C. lusitaniae, менее чем за два часа. Что касается реальных положительных КК, система смогла обнаружить 19 C. albicans, 13 C. parapsilosis, 9 C. glabrata и 2 смешанные КК C. albicans плюс C. parasilosis в полном соответствии с идентификацией с помощью масс-спектрометрии MALDI-TOF.
Выводы:
Наши результаты показывают, что молекулярный анализ с помощью картриджа lab-on chip может быть надежным и быстрым методом идентификации видов Candida в положительных КК. Дальнейшие эксперименты позволят оценить эффективность анализа и лучше определить его роль в рабочем процессе диагностической лаборатории.
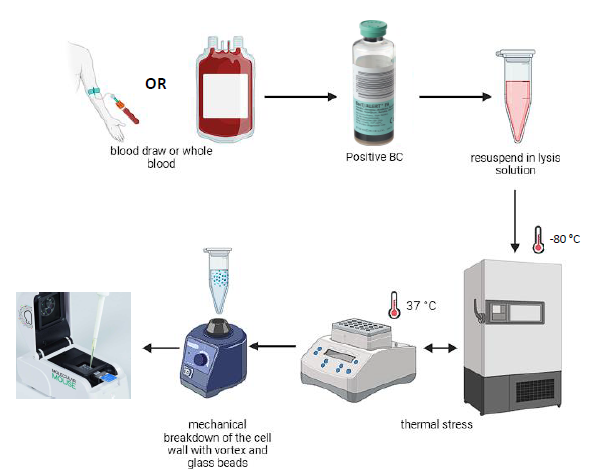
Рис. 1. Основные этапы методики выделения ДНК Candida.
Источник: Trends in Medical Mycology, 2022.
Более подробно о системе Molecular Mouse - в нашем каталоге.

