Интегративная структурная биология стремится визуализировать полную картину функционирования макромолекул, используя множество методов для просмотра молекулярных структур и их взаимодействия друг с другом.
N4-фаг из Escherichia coli и растущий список N4-подобных бактериофагов представляют собой одни из самых малоизученных бактериальных вирусов в биологии. Долгое время классифицировавшиеся как Podoviridae из-за относительно небольшого размера хвостового аппарата (на основе анализа электронной микроскопии с негативным окрашиванием низкого разрешения), N4-подобные фаги генетически и структурно значительно отличаются от классических Podoviridae.
В 2020 году масштабный биоинформационный анализ выявил 115 N4-подобных вирусов, названных Schitoviridae в честь Джан-Карло Скито, ученого, впервые выделившего фаг N4 из кишечной палочки в 1966 году из канализации в Генуе. Семейство Schitoviridae включает в себя восемь подсемейств и множество новых родов. Число N4-подобных фагов, охарактеризованных в последние годы, продолжает расти: с 33 представителей в 2015 году до 115 вирусов в 20201 году, причем за последние три года было обнаружено еще больше. Геномная сложность Schitoviridae поразительно масштабна и соответствует размеру генома ~75 кб, что почти вдвое больше, чем у большинства Podoviridae, таких как P22 или T7, которые кодируют около 90 ORF, включая три гена, кодирующие две РНК-полимеразы, один из которых - массивная вирион-ассоциированная РНК-полимераза (vRNAP), уникальная для этого семейства бактериальных вирусов.
Исследование, опубликованное в журнале Nature Communications, посвящено фагу Pseudomonas DEV, литическому фагу из рода N4-подобных. DEV входит в состав фагового коктейля CKϕ4, который уничтожает инфекции P. aeruginosa у личинок Galleria mellonella (восковой моли) и в моделях позвоночных животных. Соавтор работы Джино Чинголани из Университета Алабамы и коллеги описали полную молекулярную структуру фага DEV. Особенностью DEV является наличие внутри капсида вирион-ассоциированной РНК-полимеразы из 3 398 аминокислот, которая попадает в бактерию при заражении. Неожиданно исследование Чинголани показало, что вирион-ассоциированная РНК-полимераза является частью двигателя выброса генома, который вытаскивает ДНК фага из его головки после того, как фаг прикрепляется к поверхности бактерии Pseudomonas с помощью своих хвостовых волокон и проникает через внешнюю и внутреннюю мембраны клетки с помощью хвостовой трубки.
Семейство фагов Schitoviridae «представляет собой одни из самых малоизученных бактериальных вирусов в биологии, которые все чаще используются в фаговой терапии», - отмечает Чинголани. «Мы используем структурную биологию для расшифровки структурных элементов и картирования генных продуктов. Это очень важно, когда аминокислотная последовательность эволюционирует слишком быстро и не поддается обычному филогенетическому анализу».
Исследователи использовали локализованную реконструкцию с помощью криоэлектронной микроскопии, биохимических методов и генетических нокаутов, чтобы описать полную молекулярную архитектуру DEV, чей геном ДНК содержит 91 открытую рамку считывания, включающую гигантскую РНК-полимеразу, ассоциированную с вирионом. «Эта vRNAP является частью трехгенного оперона, сохранившегося во всех проанализированных нами геномах Schitoviridae», - пояснил Чинголани. «Мы предполагаем, что эти три белка выбрасываются в организм хозяина, образуя двигатель выброса генома, охватывающий клеточную оболочку».
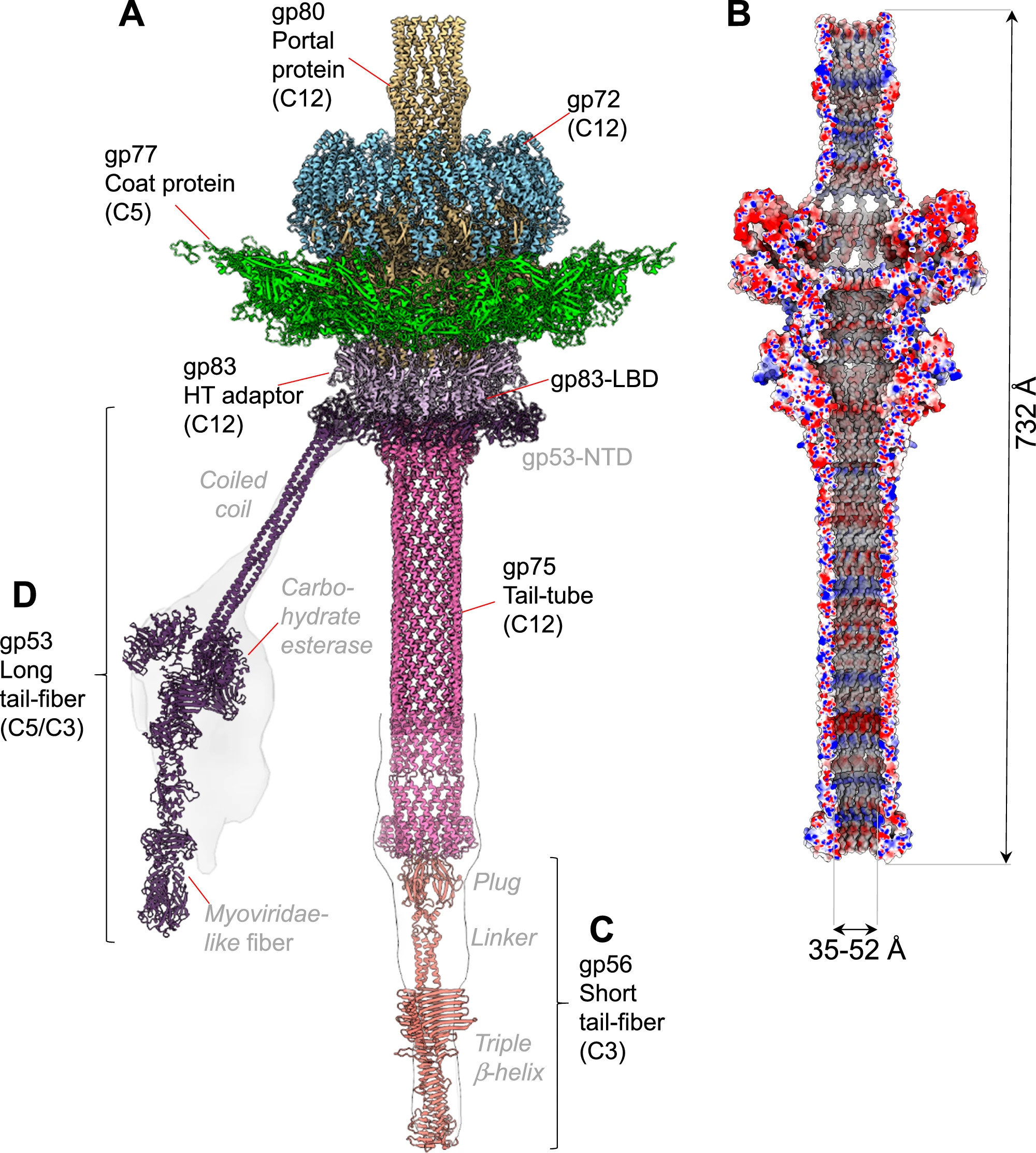
Структура DEV: большая головка или капсид, содержит геном, а волокна, похожие на ноги, поддерживают фаг, когда он приземляется на поверхность бактерии, готовясь заразить живую бактериальную клетку. Иллюстрация: Ravi K. Lokareddy et al. Nature Communications volume 15, Article number: 8482 (2024)
Исследователи определили структуры всех белковых капсидных факторов и хвостовых компонентов DEV, участвующих в прикреплении к хозяину. С помощью генетических экспериментов они показали, что длинные хвостовые волокна DEV необходимы для инфицирования P. aeruginosa, но не нужны для инфицирования мутантов P. aeruginosa, в поверхностном липополисахариде которых отсутствует О-антиген. Как правило, на первом этапе инфицирования вирусы прикрепляются к различным молекулам клеточной поверхности.
Хотя в данном исследовании представлены несколько изображений структуры фага, исследователи не до конца понимают, как происходит заражение DEV. Они предполагают три этапа этого инфекционного процесса. На первом этапе, когда один фаг DEV дрейфует в изоляции, его гибкие длинные хвостовые волокна колеблются, чтобы повысить вероятность соприкосновения с поверхностной молекулой липополисахарида Pseudomonas. После первого касания все пять волокон прикрепляются, чтобы привязать фаг перпендикулярно к внешней поверхности бактерии.
На втором этапе короткое хвостовое волокно, которое также выполняет функцию хвостовой заглушки, касается вторичного рецептора на Pseudomonas, и механический сигнал освобождает хвостовую заглушку. До этого момента три белка, называемые gp73, gp72 и gp71, хранились внутри головки фага вблизи его хвоста, и их форма резко изменится, когда они выйдут из головки фага. На третьем этапе, когда пробка исчезает, три белка выходят из головки и попадают в оболочку бактериальной клетки. Ведущий белок, gp73, меняет свою форму, образуя наружную мембранную пору с полым центром. Под ним gp72 превращается в полую трубку, которая охватывает периплазму Pseudomonas - пространство между внешней и внутренней мембранами бактерии. Наконец, gp71 пересекает внутреннюю мембрану и сворачивается в большой мотор РНК-полимеразы в цитоплазме бактерии, который протягивает фаговую ДНК через полые каналы gp73 и gp72 в клетку Pseudomonas.
«Мы предполагаем, что принципы конструкции аппарата выброса DEV сохраняются у всех фагов семейства Schitoviridae», - говорит Чинголани. «По состоянию на октябрь 2024 года более 220 геномов Schitoviridae были секвенированы и доступны в публичной базе данных. Поскольку эти геномы в значительной степени неаннотированы, а многие открытые рамки считывания имеют неизвестные функции, наша работа открывает путь для легкой идентификации структурных компонентов при обнаружении новых фагов Schitoviridae».