Исследователи из клиники King's и Guy's & St Thomas (Великобритания) обнаружили новый вид бактерий, получивший название Variovorax durovernensis.
После того как пациент, работающий пастухом в Кентербери, был госпитализирован с лихорадкой, стандартный анализ крови выявил два разных вида бактерий как источник инфекции. Однако один из видов не мог быть идентифицирован с помощью обычных лабораторных методов. Чтобы охарактеризовать этот вид, исследователи использовали нанопоровое секвенирование. Эта технология позволяет считывать длинные одиночные последовательности ДНК гораздо быстрее, чем другие методы, что дает возможность быстро проанализировать весь геном.
Результаты, опубликованные в журнале Clinical Infection in Practice, показали, что это новый род бактерий, обитающих в почве, Variovorax. Биохимический анализ и исследование генома бактерии подтвердил, что это ранее не описанный новый род. Пациент назвал новый вид Variovorax durovernensis, по латинскому названию города Кентербери, где он живет. Соавтор статьи Лара Пейн объяснила, как пациент мог заразиться: "Расследование показало, что пациент заразился во время сезона ягнения или во время кормления овец антипаразитарными препаратами, причем все это делалось без перчаток".
Наряду с новым открытием, в статье подчеркивается эффективность применения нанопорового секвенирования в клинической практике. Это быстрый и удобный метод, применяемый непосредственно в лаборатории клиники вместо трудоемкого процесса отправки образцов в специализированные лаборатории. "По мере распространения этой технологии мы, вероятно, будем открывать все новые микроорганизмы и новые инфекции", - отметила Пейн.
Авторы отмечают, что "нетипичные инфекции", вызванные микробами из окружающей среды, становятся все более распространенными, особенно среди пациентов, проходящих иммуносупрессивную терапию. Возможность легко идентифицировать необычные или новые виды бактерий в клинических лабораториях поможет быстрее выявлять инфекции и предоставлять пациентам более целенаправленное лечение.
"Мы уже используем эту технологию в клинической практике для выявления бактерий, вызывающих тяжелую пневмонию у людей, поступивших в отделение интенсивной терапии. Узнав причину инфекции, мы можем соответствующим образом подобрать антибиотики - таким образом, пациенты получают индивидуальное и целенаправленное лечение", - считает Пейн.
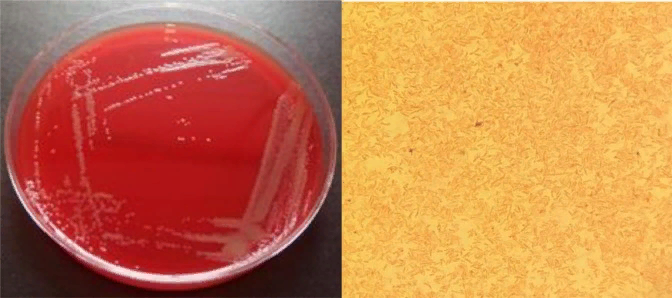
Variovorax durovernensis на шоколадном и 5% овечьем кровяном агаре; b) Грамотрицательные палочки под световым микроскопом при 100x.
Lara Payne et al. Первый зарегистрированный случай инфицирования человека Variovorax durovernensis; новый вид рода Variovorax sp., выделенный из протеза аорты пастуха (аннотация).
Мы описали случай 55-летнего овцевода с микотической аневризмой в анамнезе, у которого наблюдалась хроническая инфекция эндоваскулярного трансплантата аорты, вызванная Nocardia nova и новым идентифицированным видом Variovorax sp, который мы назвали "Variovorax durovernensis". Это первый случай, когда данный вид Variovorax sp. вызвал патологию человека. Этот организм был идентифицирован с помощью полногеномного секвенирования как новый вид Variovorax, а название "Durovernum" (латинское название Кентербери) было предложено по просьбе нашего пациента, так как это древнее название Кентербери.
История болезни
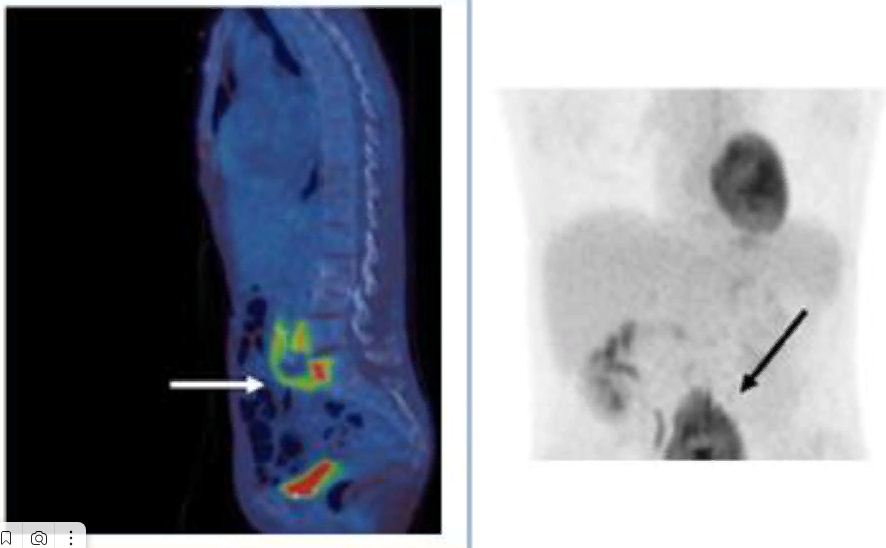
Наш пациент поступил с разрывом аневризмы левой общей подвздошной артерии с выделенным Campylobacter fetus, что потребовало экстренного эндоваскулярного стентирования. Через семь месяцев после выписки пациент повторно поступил с головной болью, потерей веса и болью в спине. Позитронно-эмиссионная томография показала инфицирование аорто-билиарного трансплантата в левой периуретеральной области. Инфицированный трансплантат был удален, и была проведена реваскуляризация с использованием аутологичных вен.
Результаты
В образцах трансплантата на 5-й день инкубации росли Nocardia nova в 3/5 образцов, а через 7 дней - желтые колонии грамотрицательного микроорганизма в 3/5 образцов. Идентификация рода Variovorax была получена с помощью масс-спектрометрии MALDI-TOF. ДНК Variovorax Sp. секвенировали на приборе GridION (Oxford Nanopore Technologies). Данные секвенирования были подвергнуты фильтрации и обработке с помощью геномных программ Minimap2 и Nanopolish v0.8.4. Epi2Me идентифицировал Variovorax sp.; геномный анализ in silico и фенотипическое тестирование in vitro подтвердили, что это новый вид.
Заключение
По мере расширения диагностических возможностей необычные клинические изоляты могут быть надежно идентифицированы. MALDI-TOF позволил идентифицировать Variovorax Sp., вызвавший клиническое заболевание в данной ситуации. Этот новый вид был определен с помощью секвенирования всего генома, что позволило идентифицировать его и признать в международных репозиториях.
Этот случай подчеркивает важность непредвзятого подхода к интерпретации результатов, полученных из клинических образцов, поскольку могут быть выделены новые нетипичные инфекции из микробиома окружающей среды.