В последние годы исследования микробиома человека показали, что дисбиоз микробных сообществ может привести к дисфункции механизмов организма хозяина, вызывая широкий спектр заболеваний.
Таким образом, понимание ассоциаций конкретных видов бактерий с заболеваниями может стать потенциальным источником новых мишеней для лечения и терапевтических подходов в клинической практике. До последних двух десятилетий традиционные методы, такие как бактериальная культура и биохимические тесты, обычно считались золотыми стандартами бактериальной диагностики и широко применялись в клинических лабораториях. Благодаря технологическому развитию и экономической выгоде, молекулярные методы, такие как ПЦР и иммуноферментный анализ, постепенно становятся доступными и популярными для диагностики бактерий.
Однако как традиционная микробиология, так и новые молекулярные методы отвечают лишь простоте и контролируемости в рамках редукционизма, фокусируясь на ограниченном количестве генов и видов бактерий. Хотя редукционистский подход может раскрыть индивидуальную генетику и физиологию, способствуя пониманию сложного поведения микроорганизмов в природе, эти наблюдения и выводы трудно напрямую применить к физиологии целых экологических систем, таких как взаимодействие человека и микробиоты.
Около десяти лет назад микробиом был всего лишь словом, о котором слышали в основном ученые, а общественность редко была знакома с этим понятием. С недавним развитием исследований микробиома все больше и больше исследователей признают, что микроорганизмы работают вместе как сообщество для достижения ключевых функций, связанных с различными аспектами здоровья человека, начиная от метаболических заболеваний, желудочно-кишечных расстройств и заканчивая эмоциональными нарушениями.
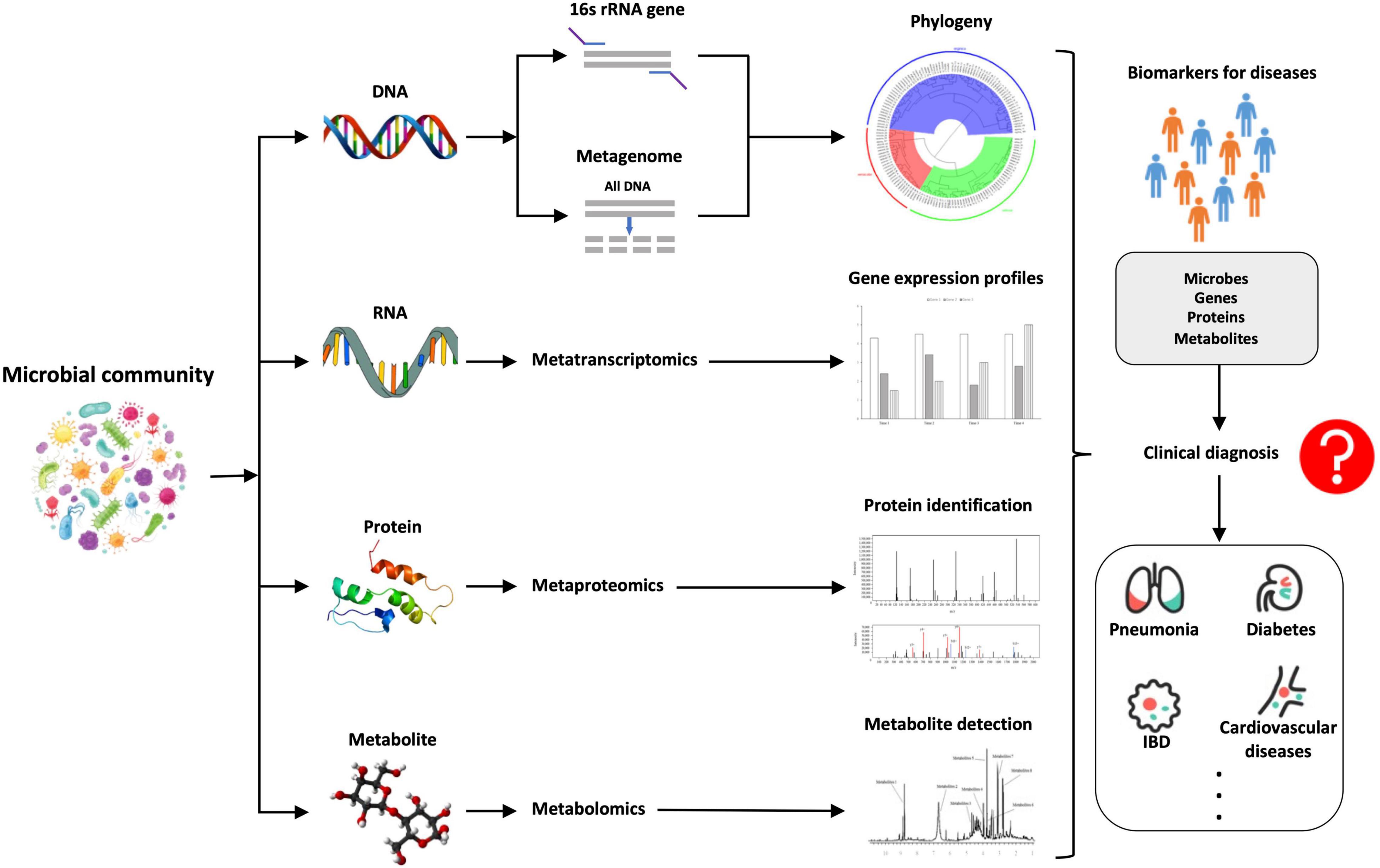
К настоящему времени разработано множество методов для пространственного и временного изучения микробных сообществ человека в таких распространенных нишах, как рот, кишечник, влагалище и т.д., которые включают метагеномику, метатранскриптомику, метапротеомику и метаболомику. Эти методы также известны как метаомика, когда их объединяют для комплексного анализа. Кроме того, общественность и популярная пресса проявляют все больший интерес к этой новой области, что закладывает основу для развития и признания метаомики в качестве инновационного инструмента бактериальной диагностики.
Метаомика - это инновационный интеграционный подход, основанный на углубленном анализе микробиомов человека, который стимулировал изменение парадигмы в понимании здоровья человека и выявлении инфекционных заболеваний. Сообщалось об очевидных преимуществах, которые делают эти методы перспективными для клинической диагностики бактериальных инфекций, таких как количественная оценка состава бактерий, обнаружение некультивируемых бактериальных патогенов, профилирование генов резистентности бактерий к антибиотикам, идентификация факторов вирулентности в больших масштабах, установление ассоциаций между бактериями и заболеваниями и т.д., и все это может быть реализовано посредством метагеномного анализа.
Кроме того, с помощью метатранскриптомных исследований можно выяснить динамику взаимодействия микробов с микробами, взаимодействия хозяина с микробами, энергетического метаболизма и химического круговорота в процессе бактериальной инфекции, что может не только улучшить понимание патогенности бактерий, но и облегчить обнаружение биомаркеров и разработку микробных терапевтических средств (Zhang et al., 2021). Более того, метатранскриптомика также способна выявлять активные бактерии и временную изменчивость экспрессии бактериальных генов во время инфекции.
Метапротеомика фокусируется на динамических изменениях целых белков в определенных микробных сообществах, что позволяет не только получить функциональную информацию о бактериальных сообществах, но и связать гены (белки) с лежащими в их основе фенотипами, что также может способствовать разработке биомаркеров для терапии и диагностики.
Метаболомика - это общий обзор метаболизма отдельных микроорганизмов, который фокусируется на глобальных профилях метаболитов (малых молекул), направленных на выявление биомаркеров для диагностики бактериальных инфекций, а также на раскрытие метаболитов, касающихся здоровья человека. Из-за сложного взаимодействия метаболитов во время взаимодействия микроба с микробом и микроба с хозяином часто строятся метаболические сети на основе реконструкции и анализа на основе ограничений (COBRA) и геномных метаболических моделей (GEM), чтобы понять связи между микробиомом и метаболомом и облегчить перевод полученных данных в эффективные и новые терапевтические средства (Heinken et al., 2021; Jansma and El Aidy, 2021).
Хотя исследования метаомики получают все более широкое распространение и предполагается, что в будущем они станут новыми диагностическими инструментами в клинической лаборатории, на современном этапе эти методы в основном ограничиваются исследовательской областью из-за недостатков, которые пока трудно преодолеть, таких как высокая стоимость экспериментальных процедур, отсутствие золотого стандарта для сбора образцов и анализа данных и т.д. Кроме того, для метаболомики методы с приемлемой чувствительностью только разрабатываются, а компьютерный анализ и интеграция данных метаомики являются другими проблемами, которые препятствуют потенциальному применению методов метаомики в клинических условиях, хотя системы управления данными и сравнительного анализа активно изучаются на современном этапе.
В данном мини-обзоре мы не будем рассматривать технические детали подходов метаомики; напротив, мы стремимся сосредоточиться на возможностях применения методов метаомики для быстрой и точной диагностики бактериальных патогенов и инфекций. Однако следует отметить, что в большинстве исследований присутствие определенных видов, измененные уровни микробов и измененное количество микробных транскриптов, белков или метаболитов не были доказаны как причины заболеваний, а только как ассоциации. Поэтому в данном мини-обзоре будут в общих чертах рассмотрены перспективы развития функциональных и трансляционных исследований микробиома в клинических условиях, которые также могут способствовать внедрению прецизионных лекарственных средств на основе метаомики.
Интегрировать данные мультиомики, например, метагеномы, метатранскриптомы, метапротеомы и метаболомы, для систематического анализа изначально сложно, поскольку эти данные в значительной степени гетерогенны и получены из разных временных шкал. В связи с важностью метаомики для всестороннего понимания микробиомов, исследования и инструменты для интеграции различных наборов мультиомических данных становятся все более доступными, что значительно облегчает развитие и трансляционный потенциал подхода метаомики в области микробиоты человека.
К настоящему времени многие пилотные исследования, предварительные анализы и комплексные исследования инновационно изучили подход метаомики для изучения микробных сообществ и их взаимодействия с хозяевами. Например, Valles-Colomer et al. (2016) провели систематический обзор применения метаомики при сложном и многофакторном воспалительном заболевании кишечника (ВЗК), который показал, что этот подход имеет большие перспективы в обеспечении понимания ВЗК, хотя интерпретация данных метаомики на нескольких уровнях была очень сложной.
Boeri et al. (2022) обобщили текущие преимущества использования метаомики для изучения взаимодействия микробиоты и хозяина в понимании эпилепсии с акцентом на сбор образцов, экстракцию и обработку данных, что может помочь в распознавании молекулярных путей и биомаркеров для связи микробиоты и эпилепсии, что приведет к разработке новых клинических методов диагностики.
Кроме того, поскольку метаомический подход требует большого объема данных, было разработано множество вычислительных инструментов и построены конвейеры для сравнительного метаомического анализа с целью расшифровки адаптации микробных сообществ и взаимодействия микробиоты и хозяина (Segata et al., 2013; Zhai et al., 2017; Sequeira et al., 2019). Однако в этой области необходимо больше компьютерных приложений, чтобы преодолеть проблемы разнообразия и гетерогенности при интеграции данных метаомики.
Фенотипы сложных микробных сообществ постоянно формируются под влиянием динамических взаимодействий между хозяевами и ассоциированной с ними микробиотой. Для того чтобы изучить всю полноту микробных функций в ходе этого процесса, необходима оптимальная и эффективная интеграция данных мультиомики, полученных с помощью метагеномики, метатранскриптомики, метапротеомики и метаболомики, что значительно улучшит наши знания о микробиоме человека и его специфической роли в состоянии здоровья и болезни человека.